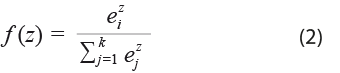Identificación de especies de plantas de la flora mexicana utilizando aprendizaje por transferencia a través de Inception-v4
Plant Species Identification of Mexican Flora Using Transfer Learning via Inception-v4
Inés Fernando Vega López,* Rito Vega Aviña,* Francisco Delgado Vargas,* Zuriel Ernesto Morales Casas,** Eduardo Díaz Gaxiola,* Juan Augusto Campos Leal,* José Abraham Berger Castro,* Gerardo Beltrán Gutiérrez* y Arturo Yee Rendón*
* Universidad Autónoma de Sinaloa (UAS), ifvega@uas.edu.mx (autor principal), rito.vega@gmail.com, fdelgado@uas.edu.mx, eduardogaxiola@uas.edu.mx, juan.campos@uas.edu.mx, abraham.bc17@gmail.com, gerardo@uas.edu.mx y arturo.yee@uas.edu.mx (autor de correspondencia), respectivamente.
** Intellion, zuriel.morales@intellion.ia
Nota: los autores desean agradecer al Consejo Nacional de Ciencia y Tecnología (CONACYT) por las becas otorgadas durante sus estudios y por el apoyo otorgado en la convocatoria del Fondo Sectorial CONACYT-INEGI 2017 en el proyecto 291772.
Vol. 14, Núm. 1 - Epub Identificación de especies... - Epub
|
A partir del 2012, las técnicas de aprendizaje profundo (AP) se han convertido en la base de grandes avances en la identificación automatizada de plantas. Este artículo presenta un estudio comparativo de arquitecturas de redes neuronales convolucionales aplicadas al problema de identificación de especies de la flora mexicana a partir de imágenes digitales. Con este fin, se construyó un modelo de clasificación a partir de la arquitectura de Inception-v4 usando un conjunto de datos de vegetación nativa de México. Este consta de 17 900 imágenes a color de 202 especies de plantas. Los resultados experimentales nos muestran que las estrategias de aprendizaje por transferencia y aumento de datos mejoran sustancialmente el desempeño de modelos basados en el AP. En particular, para Inception-v4 observamos una tasa de aciertos de 86.97 y 94.39 % en los índices Top-1 y Top-5, respectivamente. Palabras clave: técnicas de aprendizaje profundo; arquitectura CNN; identificación de especies vegetales; aprendizaje por transferencia; aumento de datos |
Since 2012, deep learning techniques have become the foundations of many breakthroughs in automated identification of plants from digital images. This paper presents a comprehensive study of Convolutional Neural Network (CNN) architectures applied to the problem of classification of Mexican plant species from digital images. For this purpose, a classification model was built based on the Inception-v4 architecture using a dataset of Mexican native flora. This dataset consists of 17 900 color images of 202 plant species. The experimental results show that transfer learning and data augmentation significantly improve a model’s performance for plant species identification. In particular, the best performance achieved by Inception-v4 was 86.97% for Top-1 accuracy and 94.39% for Top-5. Key words: Deep Learning Techniques; CNN Architecture; plant species identification; Transfer Learning; Data Augmentation. |
Recibido: 16 de marzo de 2022.
Aceptado: 22 de julio de 2022.
- Introducción
México se caracteriza por tener una amplia y rica variación geológica, orográfica y ambiental. Además, es una de las naciones con mayor diversidad biológica en el mundo. La conservación y uso sustentable de esta es indispensable para el desarrollo del país, y el impacto de su buen manejo es global.
La riqueza de especies en México está soportada en la biodiversidad de plantas que tiene; las 23 314 especies registradas al 2016 lo ubican en el cuarto lugar mundial (Villaseñor, 2016). Esta cifra aún puede aumentar de manera considerable debido a que hay extensas zonas que han sido insuficientemente exploradas o están sin explorarse, y los ecosistemas de muchas de estas zonas están siendo alterados sin tener un inventario del caudal florístico.
Conocer esta diversidad se ha vuelto una tarea complicada debido a que los trabajos de campo realizados por diferentes grupos de investigación requieren de la participación de botánicos que conozcan del manejo de las plantas y de taxónomos que las identifiquen de forma adecuada. A este respecto, el número de expertos en el país en estas materias que garanticen la identidad de las especies es muy limitado, de manera que se vuelve prácticamente imposible que cada grupo realizando trabajo de campo para hacer estudios de biodiversidad cuente con su apoyo in situ.
La idea detrás de esta investigación es demostrar que el uso de herramientas tecnológicas, basadas en la inteligencia artificial (IA), es una estrategia viable para solventar el déficit de expertos en actividades relacionadas con el estudio y la cuantificación de la biodiversidad de una región. En este sentido, la comunidad científica ha tenido algunos avances desde el 2010, sobre todo en el ámbito internacional, con el desarrollo de algunos sistemas para la identificación automatizada de plantas (Müller, 2010). Las primeras propuestas reportadas en la literatura se enfocan en la identificación de un número reducido de especies de plantas (50 o menos) a partir de imágenes de hojas tomadas en condiciones de laboratorio. Esta es una simplificación del problema que se estudia en este trabajo, pues la morfología prácticamente bidimensional de la hoja ayuda a obtener muestras fotográficas sin variaciones de escala, luz o perspectiva, por ejemplo.
Para que una propuesta tecnológica sea de utilidad práctica en estudios de diversidad florística, esta debe ser capaz de identificar, utilizando únicamente imágenes digitales, un gran número de especies. Este solo reto es muy complejo, pues se requiere lidiar con formas y texturas irregulares, además de mucha variabilidad intraclase y pequeñas diferencias interclase (Sulc et al., 2014), aunado a las variaciones morfológicas (entre órganos) y fenológicas de cada especie. Se agrega complejidad si se espera que la identificación se realice con imágenes adquiridas en condiciones de campo, pues esto incorpora factores de variación, como cambios de iluminación, color, posición, rotación, fondo y escala.
En este contexto, las técnicas tradicionales de IA y aprendizaje de máquina (AM) no logran un desempeño aceptable. Sin embargo, las de IA basadas en el aprendizaje profundo (AP, en inglés deep learning) combinadas con las de aprendizaje por transferencia (AT, en inglés transfer learning) y de aumento de datos (AD, en inglés data augmentation) han permitido la generación de propuestas de solución a este problema con resultados prometedores. Esto ha quedado reflejado en las diferentes ediciones del reto LifeCLEF (Joly et al., 2014), un evento anual destinado a impulsar la investigación en inteligencia artificial aplicada a estudios de biodiversidad a través de retos científicos en el área de aprendizaje de máquina. En particular, PlantCLEF (que forma parte de LifeCLEF) ha fomentado el desarrollo de propuestas metodológicas con resultados sobresalientes en la identificación de plantas a partir de imágenes digitales. Destacan por sus resultados el uso de redes neuronales convolucionales (CNN, por sus siglas en inglés). PlantCLEF consiste en identificar especies utilizando imágenes de uno o más de sus órganos distintivos, como hojas, frutos, tallos y flores. Inicialmente, el número de estas usado en el reto era muy limitado, con menos de 100 especies y poco más de 5 mil imágenes. Al día de hoy, ambas cantidades han crecido de manera significativa, alcanzando su máximo en el 2019 con 10 mil y 434 251, respectivamente.
Este trabajo presenta una parte de los resultados de la ejecución del Fondo Sectorial CONACYT-INEGI 2017, proyecto 291772, para generar una herramienta informática para el reconocimiento de especies vegetales, características de los tipos de vegetación de México, a partir de fotografías tomadas con dispositivos móviles. Las principales contribuciones del estudio son las siguientes: a) una evaluación exhaustiva que demuestra la efectividad de la arquitectura de red neuronal profunda, Inception-v4, para la identificación automatizada de especies de la flora nativa de México a partir de imágenes tomadas por dispositivos móviles en condiciones de campo y b) una base de datos, curada por taxónomos expertos, con imágenes de 202 especies de la vegetación del país, donde manualmente se registraron zonas de interés de los órganos de las plantas para favorecer la construcción de modelos que permitan la identificación automatizada de estas especies utilizando técnicas de aprendizaje profundo. Esta base de datos puede ser utilizada como un punto de partida para el desarrollo de nuevas y mejores técnicas de identificación de flora.
El resto de este trabajo se organiza de la siguiente manera: la segunda sección incluye una descripción de las redes neuronales convolucionales y una revisión de la literatura de trabajos basados en técnicas de AP para la identificación automatizada de plantas a partir de imágenes; la tercera presenta la metodología propuesta, en particular, se describen la arquitectura de CNN Inception-v4, el conjunto de datos, además de las técnicas de aprendizaje por transferencia y aumento de datos; la cuarta contiene la evaluación experimental y los resultados que muestran la tasa de aciertos de los modelos generados utilizando diferentes arquitecturas de redes neuronales profundas; y en la quinta se dan las conclusiones.
II Clasificación de objetos en imágenes mediante CNN
En contraste con las técnicas tradicionales de AM, por ejemplo, las máquinas de vectores de soporte y árboles de decisión, las CNN tienen la ventaja de ser capaces de encontrar por sí mismas las características visuales que ayudan a discriminar entre las clases que se les presentan; esto elimina la necesidad de desarrollar manualmente detectores de características especializados, una actividad conocida por el término ingeniería de características (feature engineering). El principal problema que se encuentra en el uso de CNN es la gran cantidad de datos que se requieren para su entrenamiento; sin embargo, proyectos como PlantCLEF han contribuido a la obtención de un conjunto de datos que cubre la alta diversidad de vegetación en el planeta. Además, en la actualidad, otros como Pl@ntNet[1] e iNaturalist[2] se apoyan de comunidades de voluntarios para obtener y clasificar plantas alrededor del mundo.
Descripción de CNN
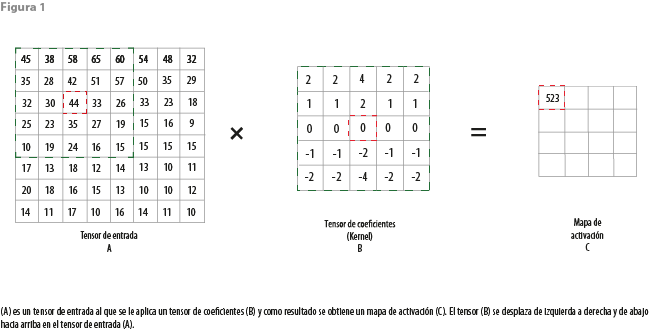
La idea básica detrás de las CNN es el uso de convoluciones. Una convolución es un operador matemático que transforma dos funciones y en una tercera que representa la magnitud en la que se superponen y una traslación invertida (reflejada) de (Kim, 2019; Fernández-Blanco, 2019). En el ámbito de las CNN, se describe como una operación matemática que recibe como entrada un tensor (arreglo multidimensional de datos) y aplica sobre él uno de coeficientes (kernel) para obtener un mapa de activación (activation map) del tensor de entrada. La convolución se realiza deslizando el kernel sobre un tensor de entrada, generalmente comenzando en la esquina superior izquierda, para moverlo a través de todas las posiciones donde se ajuste por completo dentro de los límites del tensor de entrada (Kim, 2019).
Un ejemplo del proceso de convolución se ilustra en la figura 1, donde se realiza una multiplicación elemento a elemento utilizando el tensor de entrada A y el de coeficientes B, es decir, cada elemento del primero se multiplica por el elemento correspondiente en el segundo; después, se suman los valores de las multiplicaciones y se guarda el resultado en el mapa de activación C. El área de acción del kernel está denotada con un borde verde y el elemento resultante en el mapa de activación C está en color rojo. Este proceso se aplica a todo el tensor de entrada, desplazando el kernel sobre este, como previamente hemos descrito.
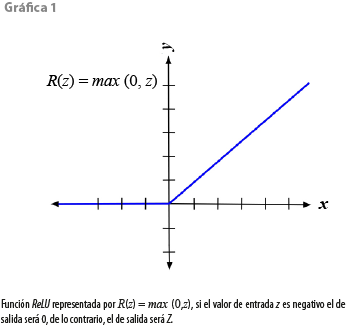
La idea principal detrás del proceso de convolución es capturar algún patrón contenido en el tensor de entrada (al principio es una imagen) y pasarlo a la siguiente capa de convolución. Los valores negativos no representan información importante en el patrón y son convertidos a 0, mientras que los positivos pasan de manera intacta a la siguiente capa. Por lo tanto, se debe definir una función que permita procesar los valores de salida del proceso. ReLU (del inglés Rectified Linear Unit), ecuación 1, es una función lineal que genera como valor de salida la entrada recibida si es positiva, y de 0 si es negativa:
R(z)=max(0,z) (1)
ReLU es la función de activación más utilizada en CNN. La gráfica 1 ilustra el comportamiento descrito de esta.
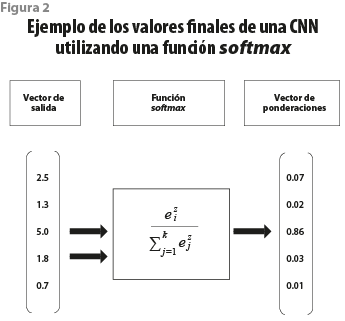
Además del operador convolución del cual toman su nombre, las CNN utilizan uno de agrupación (pooling). Una capa de agrupación en una CNN opera sobre el mapa de activación para reducir la cantidad de datos al quedarse solo con un valor representativo de una región del mapa. Esto se realiza mediante la extracción de estadísticas, por ejemplo, el promedio (average pooling) o el máximo (max pooling) de una región fija. Por último, para que los resultados de una CNN puedan ser interpretados como una probabilidad, la capa final (capa de softmax) utiliza la función softmax, también conocida como exponencial normalizada, para asignar probabilidades a cada clase en un problema de múltiples clases. La figura 2 ilustra un ejemplo del proceso para obtener las probabilidades para cada clase. Al vector de ejemplo del lado izquierdo se le aplica la función softmax, descrita en la ecuación 2, y como resultado se obtiene el de ponderaciones del lado derecho:
El rango de salida de la función softmax es [0, 1], y la suma de todos los valores en el vector de salida debe ser igual a 1.
Trabajos relacionados
Al día de hoy, las técnicas basadas en aprendizaje profundo generan los mejores resultados en el reto de identificar los objetos presentes en una imagen (He y Tian, 2016). La comunidad científica ha organizado una gran cantidad de retos en los que se invita a colegas investigadores a hacer propuestas para la clasificación de objetos en imágenes. El de ImageNet es, sin duda, el más altamente referido. Este evalúa técnicas de detección de objetos genéricos y clasificación de imágenes a gran escala. Aunque este reto se ha modificado con el tiempo, su esencia es la siguiente: dado un conjunto de imágenes se debe diseñar un modelo que permita, a una computadora, clasificar mil clases distintas de objetos en estas imágenes. Para estos retos, el comité organizador hace público un conjunto de imágenes que han sido manualmente anotadas. En el 2012, la arquitectura AlexNet (Krizhevsky et al., 2012) se convirtió en la primera propuesta de CNN en ganar el reto ImageNet. Desde entonces, las CNN se han mantenido al frente en tareas de clasificación de objetos a partir de imágenes, obteniendo resultados consistentemente superiores a cualquier otra técnica en diversos retos de clasificación.
La clasificación automatizada de organismos biológicos a partir de imágenes ha tomado gran relevancia. La de plantas, en particular, es un reto muy complejo, pues la cantidad de especies que hay es muy grande y pueden existir semejanzas significativas entre especies. Lee et al. (2015) realizaron una comparación entre un modelo de clasificación de AlexNet y métodos tradicionales basados en la extracción manual de características; este trabajo utilizó como base de comparación al conjunto de datos MalayaKew Leaf, que consiste en imágenes de hojas de 44 especies de plantas; los resultados revelan que el AlexNet superó, de manera significativa, a los métodos tradicionales mencionados. Reyes et al. (2015) utilizaron un modelo de clasificación de AlexNet y lo reentrenaron con el conjunto de datos de LifeCLEF; esta propuesta obtuvo una tasa de aciertos de 48.7 % en el reto principal de LifeCLEF 2015, superando los mejores resultados obtenidos en la edición previa del reto.
En fecha reciente, los trabajos con CNN se han enfocado en optimizar las arquitecturas de los modelos para incrementar la precisión en las predicciones y para clasificar utilizando múltiples órganos distintivos de las plantas (Lee et al., 2018). En el 2016, Lee et al. (2016) utilizaron la arquitectura VGG-16 como base de su propuesta para la clasificación de especies basada en múltiples órganos; el modelo combina capas para órganos, especies y de fusión, esto para el reto PlantCLEF 2016; el objetivo de la edición 2016 de este fue identificar mil especies vegetales y, a su vez, rechazar clases desconocidas. En el mismo año, Hang et al. (2016) utilizaron una red tipo VGG-16, la cual fue modificada reemplazando la última capa de agrupación (pooling) por una llamada spatial pyramid pooling, que da como resultado una salida de tamaño fijo; los autores también sustituyeron la función de activación ReLU por una Parametric ReLU, alcanzando un puntaje MAP (Mean Average Precision) de 0.827, el más alto de entre todos los participantes del reto. Por otro lado, Mehdipour et al. (2016) utilizaron en conjunto dos arquitecturas de redes convolucionales, GoogLeNet (ahora conocida como Inception) y VGG-16, además de usar AT del reto ImageNet; los modelos generados por esas arquitecturas fueron reentrenados con el conjunto de datos de LifeCLEF 2015; adicionalmente, los autores entrenaron un modelo de GoogLeNet para rechazar imágenes que no correspondieran a plantas en general; el sistema alcanzó una tasa de aciertos de 73.8 por ciento.
En el 2017, Toma et al. (2017) utilizaron aprendizaje por transferencia de un modelo de AlexNet para el reto de clasificación PlantCLEF 2017; los autores reportaron un MRR (mean reciprocal rank) de 0.361 en su mejor corrida. Pawara et al. (2017), por su parte, emplearon AlexNet y GoogLeNet para clasificar los conjuntos de datos Folio, AgrilPlant y Swedish Leaf usando, a su vez, una serie de técnicas de AD con la finalidad de mejorar la tasa de aciertos de los modelos. Por otro lado, Barré et al. (2017) diseñaron un sistema para la identificación de plantas basado en CNN llamado LeafNet, el cual demostró tener un desempeño superior a métodos tradicionales para la clasificación de imágenes en los conjuntos de datos de Foliage, LeafSnap y Flavia; los autores reportaron las siguientes tasas de aciertos: 95.8, 86.3 y 97.9 %, respectivamente. Carpentier et al. (2018) utilizaron la arquitectura ResNet para identificar especies de árboles nativos de Canadá a partir de sus cortezas; los autores crearon un conjunto de datos de 23 mil imágenes de estas de 23 diferentes especies: la tasa de aciertos del modelo de clasificación osciló entre 93.88 % (para varios recortes en una sola imagen) a 97.81 % (empleando todas las imágenes del tronco).
En el 2020, Zhao et al. (2020) generaron un clasificador de múltiples órganos para identificar 17 especies de árboles de la flora de China; para ello, utilizaron las arquitecturas ResNet50 y DenseNet121 como extractores de características de imágenes de órganos de corteza y hojas empleando una máquina de vectores de soporte como clasificador; el conjunto de datos usado corresponde a 400 imágenes de cada órgano por cada especie, y la tasa de aciertos reportada por los autores es de 84 % y este porcentaje se incrementa a 92 cuando se utiliza la técnica de aumento de datos. En el mismo año, Hieu et al. (2020) realizaron un análisis comparativo de diversas arquitecturas de CNN; en este trabajo crearon un conjunto de datos de la flora de Vietnam que contiene 28 046 imágenes de 109 especies, las cuales fueron recolectadas a partir de descargas automatizadas del sitio de Enciclopedia de la vida (EOL, por sus siglas en inglés); las arquitecturas comparadas fueron VGG-16, Resnet V2, InceptionResnet V2 y MobileNet V2; sus resultados experimentales indican que la tercera es la que obtiene la mejor tasa de aciertos con 83.9 por ciento.
En síntesis, el estado del arte indica que la identificación de especies de plantas es un reto muy complejo, pues se requiere lidiar con formas y texturas irregulares, además de mucha variabilidad intraclase y pequeñas diferencias interclase. Por otro lado, la disponibilidad de datos e imágenes para cada especie de la flora es diferente. Existen especies muy representadas, bien sea por su atractivo o amplia distribución geográfica, y hay otras con escasa representación, quizá por su poca frecuencia o distribución alejada de grandes asentamientos humanos. En la siguiente sección se describe la propuesta metodológica para dar solución al problema de identificación de especies de la flora nativa de México.
III. Metodología
Nuestra propuesta pretende resolver un problema denominado clasificación de grano fino. De acuerdo con la revisión de literatura, las complejidades de la identificación automatizada de especies de plantas son, al menos, las siguientes:
a) La cantidad de especies que hay es muy grande y pueden existir semejanzas significativas entre especies.
b) Muchas presentan una gran variación morfológica entre individuos. Incluso, el mismo individuo puede exhibir variaciones morfológicas y fenológicas significativas de manera estacional; por ejemplo, el paso de una flor a fruto o la maduración y caída de hojas.
c) Existen grupos florísticos, o zonas geográficas, escasamente explorados y muestreados, lo que nos enfrenta a un problema de deficiencia de datos.
La presente investigación está basada en el uso de redes neuronales convolucionales. Se evalúan varias arquitecturas para determinar aquella que nos ofrece el mejor desempeño en el caso de estudio. Las consideradas son las que han reportado los mejores resultados en tareas de identificación de especies de plantas. En concreto, se evaluó Inception-v4 y se contrastó su desempeño contra AlexNet, VGG-16 y VGG-19. Los resultados experimentales fueron ratificados con el método de validación cruzada de -iteraciones (conocido en inglés como -fold cross-validation).
Para lidiar con la complejidad derivada de la diversidad morfológica de las plantas, hemos creado un conjunto de imágenes de flora nativa de México en el que se han indicado claramente los órganos de interés de una especie, y cada uno se considera una clase diferente en el problema de clasificación. La creación de este conjunto de datos también ayuda a mitigar la complejidad derivada de la deficiencia de estos. También, para atender esa faltante, se hace uso de las estrategias de aumento de datos y de aprendizaje por transferencia.
A continuación, describimos de manera detallada la arquitectura Inception, el conjunto de datos utilizado, así como los conceptos de AT y AD.
Arquitectura Inception
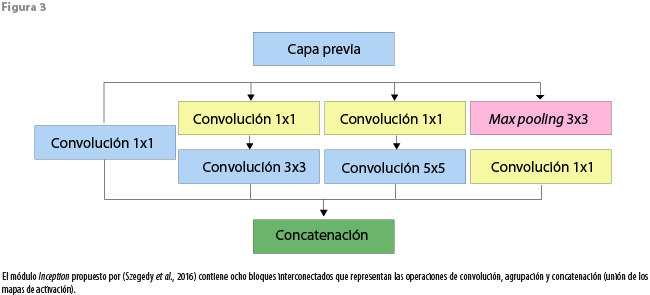
Investigadores de Google presentaron la primera versión de Inception en el 2015 bajo el nombre de GoogLeNet (Szegedy et al., 2015); en su trabajo, los investigadores señalan el problema de seleccionar un tamaño correcto del kernel debido a la potencial variación en el tamaño y la localización de las áreas de interés en las imágenes. Para solucionar esto, se propuso el módulo Inception (ver figura 3), que consiste de una combinación de capas convolucionales paralelas, cuyas salidas se concatenan al final. En concreto, cuenta con cuatro posibles rutas que van desde la capa previa hasta un nodo de concatenación. Tres de estas tienen capas convolucionales con filtros de 1 × 1, 3 × 3 y 5 × 5 y la cuarta posee una de agrupación máxima (max pooling). Todas las variantes de la arquitectura Inception consisten en diversas configuraciones de este módulo.
La principal ventaja del módulo Inception es que le permite a la red determinar el tamaño de filtro más relevante para aprender, ya que utiliza filtros de distintos tamaños en sus capas convolucionales. En particular, la arquitectura Inception-v4 es una simplificación de versiones anteriores que contiene más módulos de Inception. El número de parámetros entrenables es de 43 millones.
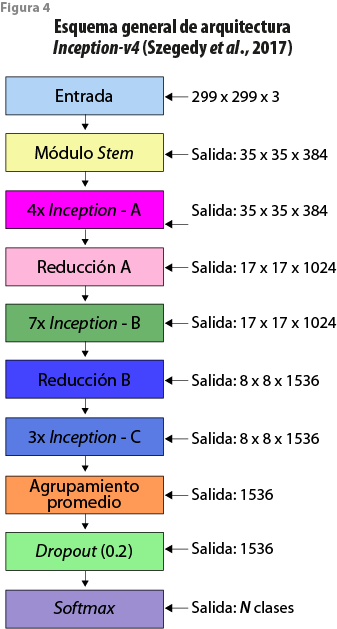
La figura 4 muestra la arquitectura de Inception-v4 (Szegedy et al., 2017), que consiste de un módulo stem, cuatro Inception A, otro de reducción A, siete de Inception B, uno de reducción B, tres de Inception C, una capa de agrupamiento, una de dropout (que permite eliminar un porcentaje de parámetros para evitar el sobreajuste del modelo) y, por último, una capa final (softmax).
El módulo Stem está compuesto por 11 capas convolucionales y dos de agrupación máxima con filtros de 1 × 1, 3 × 3 y 7 × 1. El Inception A utiliza siete capas convolucionales y una de agrupación promedio con filtros de 1 × 1 y 3 × 3. El Inception B tiene 10 convolucionales y una de agrupación promedio con filtros de 1 × 1, 1 × 7 y 7 × 1 . El Inception C contiene 10 convolucionales y una de agrupación promedio con filtros de 1 × 1, 1 × 3 y 3 × 1. El de reducción A tiene cuatro convolucionales y una de agrupación máxima con filtros de 1 × 1 y 3 × 3. El de reducción B está compuesto por seis convolucionales y una de agrupación máxima con filtros de 1 × 1, 1 × 7 y 3 × 3 y 7 × 1.
Descripción del conjunto de datos
El conjunto de datos utilizado en los experimentos que aquí se reportan fue generado principalmente a partir de imágenes tomadas durante expediciones a campo realizadas por equipos de biólogos y taxónomos expertos. Se usaron también las descargadas del sitio web de Naturalista (CONABIO, 2021), una red social en línea para compartir información sobre biodiversidad, y del reto iNaturalist (2019). Las imágenes originales tomadas de estas tres fuentes pasaron por una etapa de preprocesamiento manual para indicar regiones de interés. Cada una de las (anotación de órganos distintivos) marcadas en las imágenes originales generó una nueva imagen que se incorporó al conjunto de datos utilizado para construir y validar los modelos. Algunos ejemplos de anotación se muestran en la figura 5.
Las regiones marcadas se extrajeron de las imágenes originales para generar una nueva por cada área de interés. Las generadas en este proceso son siempre cuadradas. Estas se anotaron y recortaron cuidadosamente mediante el uso de una herramienta desarrollada por los autores, denominada PitCrop.[3] El número de imágenes incorporadas en este conjunto de datos a partir de las expediciones fue de 11 250 y en ellas se representan 139 especies de plantas. Del sitio de Naturalista se tomaron e incorporaron 4 350 de 46 especies, mientras que del reto iNaturalist se incluyeron 2 300 de 17 especies. En total, el conjunto de datos consta de 17 900 imágenes en color de 202 especies de plantas de la flora mexicana, las cuales se dividen en 358 clases con 50 imágenes cada una y que corresponden a órganos distintivos de las especies, como hojas, flores, tallos y frutos, aunque no todas tienen representación de todos sus órganos en este conjunto de datos. Además, las imágenes del conjunto final contienen variaciones en el fondo, ángulos, iluminación, contraste y escala. Algunos ejemplos se presentan en la figura 6.
Aprendizaje por transferencia
Este permite reutilizar los valores de los parámetros adquiridos por un modelo de clasificación. La idea principal de esta técnica es utilizar el conocimiento obtenido de un modelo entrenado en una tarea y aplicarlo a una segunda similar. En la práctica, se emplean modelos que fueron entrenados empleando conjuntos de datos a gran escala, como ImageNET y PlantCLEF. Debemos destacar que, en el aprendizaje por transferencia, solo se usan los valores de los parámetros (coeficientes) de las capas convolucionales del modelo base (previamente entrenado). Por lo común, hay dos formas de utilizar los modelos previamente entrenados. La primera es conocida como extracción de características (feature extraction) y la segunda, como refinamiento (fine-tuning). Cuando un modelo se usa como un extractor de características, los valores de los parámetros de las capas convolucionales no se modifican; por otro lado, en el refinamiento se utilizan los del modelo base como punto de partida y se ajustan estos valores durante el entrenamiento del modelo en otro conjunto de datos destino. Los principales beneficios del AT son la reducción del tiempo de entrenamiento de los modelos y la mejora del desempeño del modelo final. Además, esta técnica permite mitigar la ausencia de una gran cantidad de datos para el entrenamiento de los modelos (Torrey y Shavlik, 2009).
Aumento de datos
Cuando se busca generar modelos basados en aprendizaje profundo, la disponibilidad de datos es siempre un problema (y no menor). La cantidad de parámetros entrenables es muy grande (millones) y si el conjunto de datos no tiene suficientes observaciones, ocurrirá un sobreentrenamiento. Cuando no es posible conseguir más datos, se recurre a una estrategia llamada AD, que consiste en generar nuevas observaciones aplicando transformaciones a las imágenes existentes, como rotación, reflexión y escalamiento.
En la siguiente sección se describen las configuraciones experimentales que se realizaron para encontrar el mejor modelo de clasificación de especies de la flora nativa de México. Para ello, se evaluaron cuatro arquitecturas de redes neuronales profundas con técnicas de aprendizaje por transferencia y aumento de datos.
IV. Experimentos y resultados
Los experimentos fueron realizados en una computadora especializada con las siguientes características de hardware/software: procesador Intel Xeon W-2133, 32 GB de RAM, tarjeta de procesamiento gráfico NVIDIA GTX 1080, sistema operativo Ubuntu 18.04, CUDA toolkit 10.0. Para la implementación de las arquitecturas de redes neuronales profundas se utilizó Keras versión 2.2.4, que es una interfaz de programación de aplicaciones orientada al diseño y manejo de modelos de aprendizaje máquina, escrita en Python. Además, se usó la biblioteca de software TensorFlow 1.13.1 y se creó un entorno virtual de ejecución con Anaconda 4.6.7.
Consideraciones generales para el entrenamiento de los modelos
Para validar los modelos basados en CNN, se empleó el método de validación cruzada de -iteraciones (conocido en inglés como -fold cross-validation). Este consiste en dividir el conjunto de datos de forma aleatoria en subconjuntos de aproximadamente el mismo tamaño; se utilizan subconjuntos para entrenar el modelo, y con el subconjunto restante se valida. Este proceso se repite veces usando un subconjunto distinto como validación en cada iteración. Este método genera estimaciones del desempeño, cuyo promedio se emplea como estimación final. Para este trabajo, se utilizó un valor de .
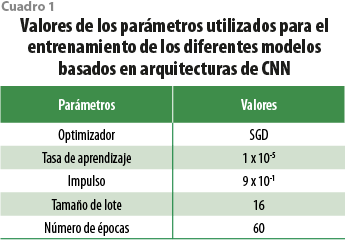
En el entrenamiento de los modelos de clasificación, se empleó el algoritmo de entrenamiento de descenso de gradiente estocástico (SGD, por sus siglas en inglés) con una tasa de aprendizaje de , impulso (momentum) de , tamaño de lote de 16 y 60 épocas. Como función de pérdida se utilizó la entropía cruzada categórica. El cuadro 1 proporciona un resumen de los valores de los parámetros usados durante el entrenamiento de los modelos.
Los resultados experimentales fueron reportados como la tasa de aciertos en las primeras respuestas (conocido en inglés como Top- accuracy); principalmente, se reportan el Top-1, es decir, la respuesta del modelo debe ser exactamente la esperada, y el Top-5, que significa que cualquiera de las cinco primeras respuestas de mayor probabilidad del modelo debe coincidir con la esperada.
Descripción de los experimentos
Los experimentos se dividieron en tres etapas. En la primera se entrenan los modelos en evaluación sin utilizar las estrategias de aprendizaje por transferencia ni aumento de datos; esto nos ayuda a establecer una línea base del desempeño de los modelos. La segunda consiste en usar como base modelos de clasificación previamente entrenados en el reto de ImageNet para hacer AT. En la tercera se emplean los entrenados en el reto de PlantCLEF 2018. En este caso solo se utiliza la arquitectura que obtuvo la mejor tasa de aciertos en la segunda. Para las etapas segunda y tercera, las configuraciones experimentales para el entrenamiento y comparación de los modelos de clasificación son las siguientes:
- Entrenamiento de modelos utilizando solo la técnica de aprendizaje por transferencia de modelos previamente entrenados haciendo un reentrenamiento completo.
- Entrenamiento usando las técnicas de AT (reentrenamiento completo) y aumento de datos. Para el AD, se utilizaron tres transformaciones a las imágenes: rotación de 90⁰, así como volteo horizontal y vertical.
Resultados de la primera etapa experimental
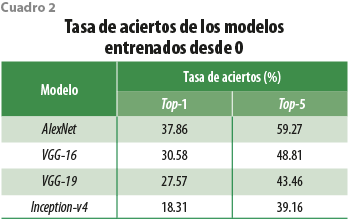
Los obtenidos en esta fase se muestran en el cuadro 2. Esos resultados son una línea base para la evaluación de los modelos en las etapas posteriores. El entrenamiento de todos se realizó a partir de valores aleatorios en sus coeficientes (se le llama entrenamiento a partir de 0). Como se puede apreciar, todos los valores son pobres (inferiores a 40 % en tasa de aciertos para el índice Top-1), aunque se aprecia que los modelos más simples obtienen mejores resultados. Los valores bajos son de esperarse, pues el número de imágenes en el conjunto de datos utilizado para entrenar los modelos es muy pequeño. Todas las arquitecturas evaluadas fueron propuestas para clasificar las imágenes del reto ImageNet, cuyo conjunto de datos contiene 1 millón de estas. Las 17 900 imágenes en el conjunto de datos empleado en este experimento simplemente resultan insuficientes para que los modelos obtengan una mayor tasa de aciertos.
Resultados de la segunda etapa experimental: modelos basados en ImageNet
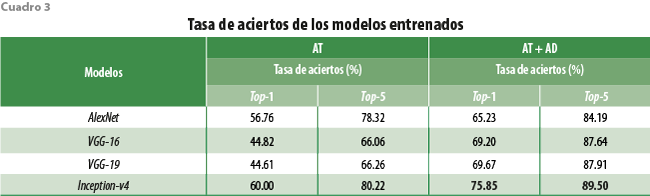
Los obtenidos en esta fase se muestran en el cuadro 3. Estos resultados son considerablemente mejores a los mostrados el cuadro 2 y esto se debe a lo siguiente: cuando empleamos aprendizaje por transferencia, los modelos utilizan el conocimiento adquirido de otro problema (en esta etapa experimental, del reto ImageNet) como punto de partida, y durante el entrenamiento se modifican de manera sutil sus parámetros para obtener un mejor resultado (mejor generalización) sobre el conjunto final. La mejora obtenida fue entre 14 y 42 % con respecto a los modelos entrenados desde 0. En particular, Inception-v4 obtuvo la mejor tasa de aciertos, que fue 60 % en Top-1 y 80.22 % en Top-5, respectivamente. Por otro lado, el aumento de datos permitió incrementar el conjunto de imágenes de entrenamiento y así agregar una mayor variabilidad reduciendo el sobreajuste de los modelos. Esta técnica mejoró la tasa de aciertos entre 10 y 15 puntos porcentuales sobre los modelos entrenados con aprendizaje por transferencia. Nuevamente, el mejor fue Inception-v4 con una tasa de aciertos de 75.85 % en Top-1 y 89.50 % en Top-5.
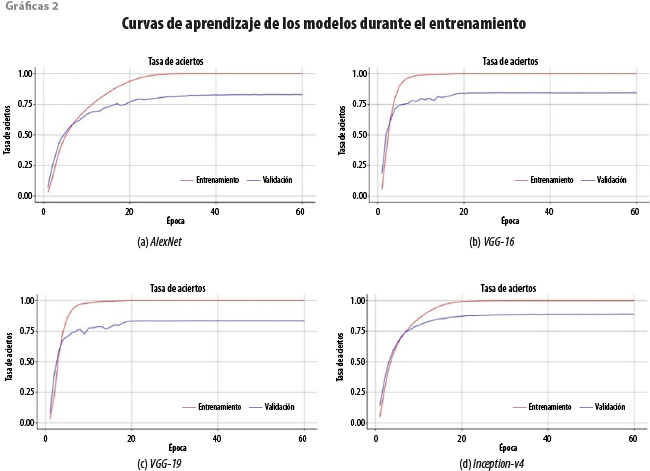
Las curvas de aprendizaje permiten conocer el comportamiento de los modelos durante el entrenamiento utilizando un conjunto de validación para medir qué tan bien están generalizando. Las gráficas 2 muestran las curvas de aprendizaje de los modelos que se evaluaron. Por cuestiones de espacio, solo se presenta una gráfica por cada arquitectura. En a, b y c, correspondientes a AlexNet, VGG-16, y VGG-19, se presenta un amplio margen entre la tasa de aciertos en el conjunto de entrenamiento (la curva color rojo) y en el de validación (la púrpura). La diferencia entre ambas curvas es cercana a 20 %, lo que sugiere un sobreajuste en los modelos. Por otro lado, la gráfica d, que es la del Inception-v4 muestra una menor diferencia (menos de 10 %), lo cual sugiere un mejor ajuste del modelo. Este análisis nos ayuda a explicar los resultados que aparecen en el cuadro 3.
Resultados de la tercera etapa experimental: modelos basados en PlantCLEF 2018
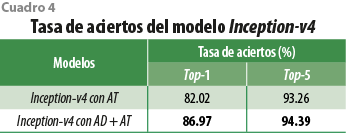
A partir de los resultados de la etapa previa, se decidió utilizar la arquitectura Inception-v4 para generar modelos que usen aprendizaje por transferencia a partir del reto PlantCLEF 2018 (es para la identificación de especies de plantas)[4] (Sulc et al., 2018). Lo obtenido se muestra en el cuadro 4 y proporciona evidencia de que emplear AT de una tarea similar a la que se está atendiendo permite generar modelos con mejores resultados que los que se generan cuando se utiliza aprendizaje por transferencia a partir de una tarea genérica (como lo es el reto ImageNet). En particular, observamos un incremento de 22 puntos porcentuales (Top-1) cuando pasamos del AT a partir de ImageNet a aprendizaje por transferencia desde PlantCLEF. Por otra parte, el modelo Inception-v4 entrenado con AT y AD fue el que obtuvo la mejor tasa de aciertos, con 86.97 % en Top-1 y 94.39 % en Top-5. Este resultado era esperado a partir de la variabilidad que las transformaciones de aumento de datos incorporan a las imágenes utilizadas para el entrenamiento de los modelos.
V. Conclusiones
La identificación de plantas es un reto complejo, aun para personas con conocimientos especializados en el área. Se trata de una tarea de clasificación donde detalles finos hacen la diferencia entre una especie y otra, mientras que, al mismo tiempo, existen discrepancias importantes entre individuos de una misma especie.
Al día de hoy, la literatura científica nos indica que las técnicas de inteligencia artificial basadas en el aprendizaje profundo son las más efectivas para automatizar esta tarea. Durante su desarrollo, los sistemas basados en estas técnicas deben exponerse a un conjunto de imágenes debidamente identificadas en un proceso conocido como entrenamiento. A mayor diversidad de especies y cantidad de observaciones por especie en este conjunto de imágenes, se espera que mejor sea el desempeño de dichos sistemas.
En la práctica, uno de los problemas para el desarrollo de sistemas para la identificación automatizada de plantas es la carencia de estos conjuntos de imágenes donde se encuentre representado un número significativo de especies de una región en particular, como en el caso de México.
En el presente trabajo hemos evaluado varias arquitecturas de redes neuronales profundas en combinación con técnicas paliativas para atender este problema de deficiencia de datos, además de crear un conjunto con imágenes de especies nativas de México. A partir de nuestra evaluación experimental, podemos concluir lo siguiente:
- La estrategia de aumento de datos durante el entrenamiento de redes neuronales profundas brinda mejoras significativas en la tarea de clasificación de plantas. Esto se cumple para las cuatro arquitecturas evaluadas con ganancias de entre 9 y 25 % en la tasa de aciertos Top-1. Ello se debe a que la estrategia de AD introduce variabilidad en el conjunto de imágenes de entrenamiento de los modelos reduciendo el sobreajuste.
- La estrategia de aprendizaje por transferencia también incorpora mejoras en el desempeño del modelo; aquí se observan ganancias en la tasa de aciertos que van desde 14 hasta 42 % si contrastamos los resultados de la línea base con respecto a los de la segunda etapa experimental. Debemos mencionar que es de gran importancia el tipo de imágenes con las que fue entrenado el modelo a partir del cual se hace la transferencia. En nuestros experimentos pudimos observar un incremento en la tasa de aciertos de aproximadamente 22 % cuando se utiliza AT desde un modelo entrenado con imágenes de plantas con respecto a partir de uno con imágenes de objetos genéricos. Por otro lado, debemos dejar en claro que, además de la limitación intrínseca de su dependencia de un conjunto de imágenes suficientemente expresivo, las técnicas de aprendizaje profundo solo pueden utilizar las características visuales presentes en una imagen durante el proceso de aprendizaje e identificación de especies.
- El uso de otras características importantes disponibles para un humano a través de sentidos como el olfato o el tacto no es posible para las técnicas de inteligencia artificial descritas en este trabajo, lo que reduce (al menos de manera potencial) su capacidad de discernir entre especies similares visualmente.
_________
Fuentes
Barré, P., B. C. Stöver, K. F. Müller y V. Steinhage. “Leafnet: A computer vision system for automatic plant species identification”, en: Ecological Informatics. 40, 2017, pp. 50-56.
Carpentier, M., P. Giguere y J. Gaudreault. “Tree species identification from bark images using convolutional neural networks”, en: Proceedings of the International Conference on Intelligent Robots and Systems. October 1-5. Madrid, España, IEEE, 2018, pp. 1075-1081.
Comisión Nacional para el Conocimiento y Uso de la Biodiversidad (CONABIO). Naturalista. 2021 (DE) http://www.naturalista.mx, consultado el 3 de diciembre de 2021.
Fernández-Blanco, R. “Deep learning para la generación de imágenes histopatológicas realistas mediante aritmética de vectores conceptuales”, en: Tesis de maestría en Bioinformática y Bioestadística. Universitat Oberta de Catalunya (UOC), 2019.
Hang, S. T., A. Tatsuma y M. Aono. “Bluefield (kde tut) at lifeclef 2016 plant identification task”, en: Proceedings of the Conference and Labs of the Evaluation Forum. September 5-8. Évora, Portugal, CEUR-WS, 2016, pp. 459-468.
He, A. y X. Tian. "Multi-organ plant identification with multi-column deep convolutional neural networks", en: Proceedings of the International Conference on Systems, Man, and Cybernetics. October 9-12. Budapest, Hungary, IEEE, 2016, pp. 2020-2025.
Hieu, N. V. y N. L. H. Hien. “Automatic Plant Image Identification of Vietnamese species using Deep Learning Models”, en: International Journal of Engineering Trends and Technology. 68(4), 2020, pp. 25-31.
iNaturalist reto 2019. iNaturalist.org. 2019 (DE) bit.ly/3uTFK85, consultado el 3 de diciembre de 2021.
Joly, A., H. Goëau, H. Glotin, C. Spampinato, P. Bonnet, W. Vellinga, R. Planqué, A. Rauber, R. B. Fisher y H Müller. “LifeCLEF 2014: Multimedia Life Species Identification Challenges”, en: Proceedings of the International Conference of the CLEF Initiative. September 15-18. Sheffield, UK, Springer, 2014, pp. 229-249.
Kim, H. “The definition of convolution in deep learning by using matrix”, en: Journal of Engineering and Applied Sciences. 14, 2019, pp. 2272-2275.
Krizhevsky A., I. Sutskever y G. E. Hinton. “Imagenet classification with deep convolutional neural networks”, en: Proceedings of the Annual Conference on Neural Information Processing Systems. December 3-6. Lake Tahoe, Nevada, United States, 2012, pp. 1106-1114.
Mehdipour, M., B. Yanıkoğlu y E. Aptoula, “Open-set plant identification using an ensemble of deep convolutional neural networks”, en: Proceedings of the Conference and Labs of the Evaluation Forum. September 5-8. Évora, Portugal, CEUR-WS, 2016, pp. 518-524.
Müller, H., P. Clough, T. Deselaers y B. Caputo. “Experimental Evaluation in Visual Information Retrieval”, en: The Information Retrieval Series. 32, Springer, 2010.
Lee, S. H., C. S. Chan, P. Wilkin y P. Remagnino. “Deep-plant: Plant identification with convolutional neural networks”, en: Proceedings of the International Conference on Image Processing. September 27-30. Quebec City, QC, Canada, IEEE, 2015, pp. 452-456.
Lee, S. H., Y. L. Chang, C. S. Chan y P. Remagnino. “Plant identification system based on a convolutional neural network for the lifeclef 2016 plant classification task”, en: Proceedings of the Conference and Labs of the Evaluation Forum. September 5-8. Évora, Portugal, CEUR-WS, 2016, pp. 502-510.
Lee, S. H., C. S. Chan y P. Remagnino. “Multi-organ plant classification based on convolutional and recurrent neural networks”. en: IEEE Transactions on Image Processing. 27(9), 2018, pp. 4287-4301.
Pawara, P., E. Okafor, L. Schomaker y M. Wiering. “Data augmentation for plant classification”, en: Proceedings of the International Conference on Advanced Concepts for Intelligent Vision Systems. September 18-21. Antwerp, Belgium, Springer, 2017, pp. 615-626.
Reyes, A. K., J. C. Caicedo y J. E. Camargo. “Fine-tuning deep convolutional networks for plant recognition”, en: Proceedings of the Conference and Labs of the Evaluation forum. September 8-11. Toulouse, France, CEUR-WS, 2015.
Sulc, M. y J. Matas. “Texture-based leaf identification”, en: Proceedings of the European Conference on Computer Vision. September 6-7 and 12. Zurich, Switzerland, Springer, 2014, vol. 8928, pp. 185-200.
Sulc, M., L. Picek y J. Matas. “Plant recognition by Inception networks with test-time class prior estimation”, en: Proceedings of the Conference and Labs of the Evaluation Forum. September 10-14. Avignon, France, CEUR-WS, 2018, p. 2125.
Szegedy, C., W. Liu, Y. Jia, P. Sermanet, S. Reed, D. Anguelov, D. Erhan, V. Vanhoucke y A. Rabinovich. “Going deeper with convolutions”, en: Proceedings of the Conference on Computer Vision and Pattern Recognition. June 7-12. Boston, MA, USA, IEEE, 2015, pp. 1-9.
Szegedy, C., V. Vanhoucke, S. Ioffe, J. Shlens y Z. Wojna. “Rethinking the inception architecture for computer vision”, en: Proceedings of the Conference on Computer Vision and Pattern Recognition. June 27-30. Las Vegas, NV, USA, IEEE, 2016, pp. 2818-2826.
Szegedy, C., S. Ioffe, V. Vanhoucke y A. Alemi. “Inception-v4, Inception-ResNet and the Impact of Residual Connections on Learning”, en: Proceedings of the AAAI Conference on Artificial Intelligence. 31(1), 2017 (DE) https://doi.org/10.1609/aaai.v31i1.11231.
Toma, A., L. Stefan y B. Ionescu. “Upb hes so @ plantclef 2017: Automatic plant image identification using transfer learning via convolutional neural networks”, en: Conference and Labs of the Evaluation Forum. September 11-14. Dublin, Ireland, CEUR-WS, 2017, p. 1866.
Torrey, L. y J. Shavlik. “Transfer Learning”, en: Handbook of Research on Machine Learning Applications. 2009.
Villaseñor, J. L. “Checklist of the native vascular plants of Mexico”, en: Revista Mexicana de Biodiversidad. 87(3), 2016, pp. 559-902.
Zhao, Y., X. Gao, J. Hu, Z. Chen y Z. Chen. “Tree species identification based on the fusion of bark and leaves”, en: Mathematical Biosciences and Engineering. 17(4), 2020, pp. 4018-4033.
[1]Pl@ntNet bit.ly/3FSTHJI
[2] iNaturalist bit.ly/3VZQDRS
[3] PitCrop está disponible al público en bit.ly/3Fwc4CX